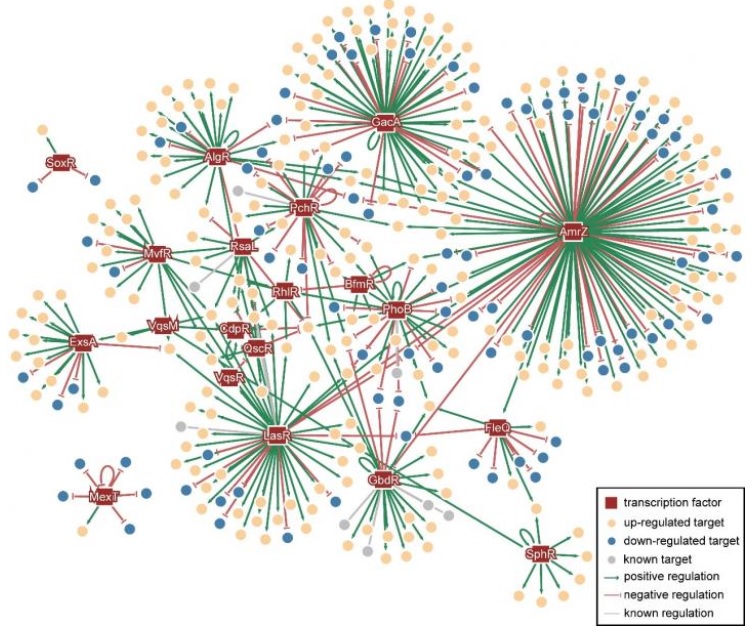
En décryptant le mécanisme régulateur de la « supervirulence » de certaines souches de Pseudomonas aeruginosa, cette équipe de l’Université de Hong Kong identifie de nouvelles protéines cibles qui pourront donner lieu au développement de nouveaux antibiotiques. Des travaux présentés dans la revue Nature Communications, qui « dessinent » un réseau nommé PAGnet constitué d’une vingtaine de régulateurs de facteurs de transcription et qui détermine la virulence de la bactérie.
Alors que la résistance aux antibiotiques est une menace de Santé publique de plus en plus préoccupante, le développement de nouveaux antibiotiques devient une urgence. L’équipe de Hong Kong révèle ici le mécanisme de régulation de la virulence de Pseudomonas aeruginosa, une superbactérie fréquente chez les patients immunodéficients et résistante à de nombreux antibiotiques. Les résultats qui ouvrent la voie à l’identification de nouvelles cibles pour de nouveaux antibiotiques. Car Pseudomonas aeruginosa est un pathogène courant en cause dans les infections nosocomiales, et résistant à l'ampicilline, l'amoxicilline et la vancomycine. Selon l'Organisation mondiale de la santé (OMS), Pseudomonas aeruginosa fait partie des 3 « agents pathogènes prioritaires » à éradiquer.
Inhiber les agents régulateurs de la virulence
Les biologistes et les informaticiens ont décrypté le réseau de régulation génomique de la bactérie et sont parvenus à identifier les régulateurs maîtres sur les principales voies pathogènes. La pathogénicité bactérienne apparaît en effet médiée par des réseaux de régulation incluant ces facteurs de transcription liés à la virulence. Ces facteurs transcriptionnels (TF) sont en fait des protéines capables d'activer et de désactiver des gènes spécifiques et constituent donc un facteur déterminant du fonctionnement du gène à un moment donné. Les régulateurs « maîtres » sont les facteurs de transcription contrôlant la plupart des activités de régulation d'autres facteurs de transcription et des gènes associés. Ces régulateurs principaux apparaissent ainsi comme des cibles prometteuses pour de nouveaux antibiotiques.
Dans le cas de Pseudomonas aeruginosa, de nombreux TF régulent la virulence. Pour découvrir de nouvelles cibles médicamenteuses contre Pseudomonas aeruginosa, les scientifiques ont cartographié tout le réseau de régulation génomique de Pseudomonas aeruginosa (PAGnet : voir visuel). Cette cartographie révèle un mécanisme de régulation de la virulence complexe, assuré par des facteurs de transcription dont 9 régulateurs maîtres.
Un progiciel basé sur PAGnet permet aujourd’hui aux chercheurs de visualiser le réseau, ses sous-réseaux, de l’explorer en vue de l’identification des TF clés impliqués dans le processus biologique de Pseudomonas aeruginosa.
Il s’agira donc maintenant de développer des inhibiteurs de ces régulateurs maîtres de la virulence bactérienne : « Les principaux régulateurs que nous avons identifiés sont des cibles antibiotiques possibles qui ont une importance clinique importante pour le développement de nouveaux antibiotiques contre Pseudomonas aeruginosa ».
Mais, au-delà, la plateforme de recherche développée va permettre de travailler à la compréhension de la résistance et de la virulence d’autres bactéries pathogènes.
Source: Nature Communications 03 July 2019 An integrated genomic regulatory network of virulence-related transcriptional factors in Pseudomonas aeruginosa (Visuel City University of Hong Kong)
Plus sur l’Antibiorésistance


