De nombreux gènes capables d'équiper les bactéries de résistance aux antibiotiques ont été identifiés dans de nombreux environnements. En répertoriant les gènes de résistance identifiés chez les pathogènes bactériens humains communs et en étant évaluant, par type de pathogène, les niveaux de risque de transfert de gènes de résistance, ces chercheurs de l’Université technique du Danemark, avec le soutien du Centre de biosécurité de la Fondation Novo Nordisk, vont pouvoir prédire pour chaque pathogène ou maladie l'évolution de la résistance aux antibiotiques et guider aussi le développement de nouveaux antibiotiques. Ces avancées, présentées dans la revue Nature Communications, devraient permettre non seulement d’ouvrir les bonnes pistes pour les antimicrobiens du futur mais aussi à terme, sur un plan clinique, aider les médecins à « mieux prescrire ».
Il s’agit, précisément, d’un large recensement des informations phénotypiques et détaillées d’une large collection de gènes de résistance. Ces informations sont cruciales alors que les bactéries sont des créatures sociales qui partagent constamment leurs gènes les unes avec les autres, que cet échange de gènes contribue à l’émergence des résistances aux antibiotiques, ces transferts pouvant transformer des bactéries inoffensives en « superbactéries ».
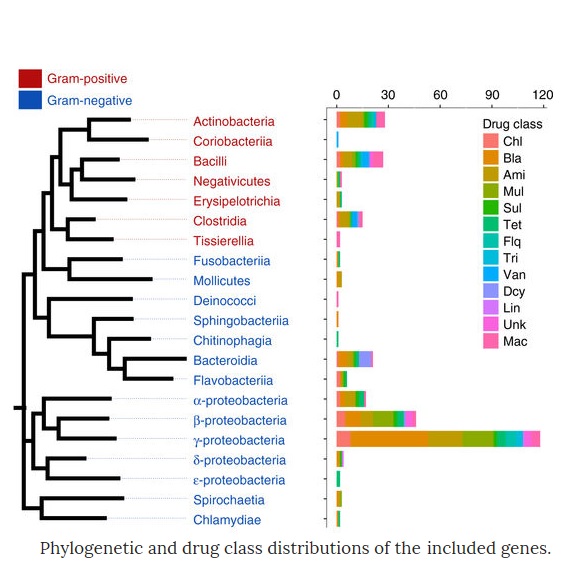
Cerner la capacité de transfert des gènes de résistance : c’est la biologie synthétique qui est à l’œuvre ici pour échantillonner une grande diversité de gènes de résistance et étudier la probabilité et l'impact du transfert de gènes sur un nouvel hôte bactérien. A partir de ces données, il devient en effet évident que le mécanisme biochimique d'un produit génique et sa compatibilité avec la physiologie de l'hôte pathogène est important dans l’émergence possible de résistances : en particulier lorsqu’il s’agit de développer de nouveaux antibiotiques afin d’éviter les molécules susceptibles d'être ciblées par des mécanismes de résistance qui peuvent facilement se disséminer parmi les hôtes pathogènes. L’exemple est donné avec les gènes responsables de la résistance à la pénicilline qui « pénètrent » facilement de nouveaux hôtes pathogènes par rapport au gènes impliqués dans la résistance à la tétracycline.
200 gènes ont ainsi été échantillonnés et l’analyse permet d’identifier les gènes les plus faciles à intégrer ou transférable à une autre cellule. Tout se résume au mécanisme biochimique et à l'hôte dans lequel les gènes sont naturellement présents.
« En biotechnologie, nous souhaitons souvent prendre des gènes d'un organisme et les exprimer dans un autre organisme, parfois lointain, afin d'obtenir de nouvelles fonctionnalités chez un autre hôte », expliquent les auteurs. Ce mécanisme est vrai également pour les pathogènes.
Mais au-delà, ces travaux pourraient contribuer à prédire les effets de différents gènes dans de nombreux autres contextes cellulaires.
Source: Nature Communications 06 February 2018 doi:10.1038/s41467-018-02944-3 Biochemical mechanisms determine the functional compatibility of heterologous genes (Visuel Christian Als / Fondation Novo Nordisk Centre pour la Biosécurité)
Plus sur l’Antibiorésistance


